FastKing RT-kit (met gDNase)
Functies
■ Hoge efficiëntie: het FastKing RT-enzym is gemodificeerd met een hydrofoob motief, met een RT-efficiëntie van meer dan 95%.
■ Gevoelig: sjablonen van slechts 1 ng kunnen nauwkeurig worden geïdentificeerd.
■ Weerstand: geschikt voor reverse transcriptie van complexe sjablonen, met perfecte weerstand tegen onzuiverheden.
■ Flexibel: verwijdering van genomisch DNA en reverse transcriptie werden afzonderlijk voltooid. Primers werden afzonderlijk in een buis gemengd, flexibel om andere primers te vervangen.
Specificatie
Type: gen-gemodificeerde reverse transcriptase, gDNase
Procedures: Tweestaps (verwijdering van genomisch DNA en RT)
RT-efficiëntie: >95%
Sjabloon: 1 ng- 2 μg
Operatie tijd: ~21 min
Toepassingen: Het omgekeerd getranscribeerde cDNA kan worden gebruikt in conventionele PCR, real-time PCR, cDNA-bibliotheekconstructie.
21 min reactie in één buis
Het duurt slechts 21 minuten om de gDNA-verwijdering en het efficiënte reverse transcriptieproces in dezelfde buis te voltooien zonder de reactiebuis en het onafhankelijke DNase I-behandelingsproces te vervangen. Vergeleken met de traditionele methode die een 12-staps werking en een reactie van 140 minuten vereist, vereenvoudigt het de bedieningsstappen aanzienlijk en bespaart het veel bedieningstijd.
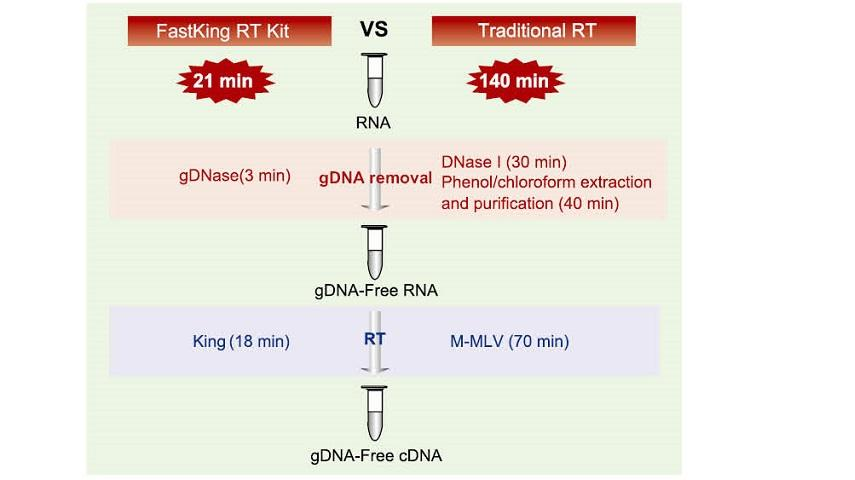
Uitstekende kwaliteit van King RTase
——Ultrahoge efficiëntie van omgekeerde transcriptie
——Reverse transcriptie efficiëntie is meer dan 95%
De algemene reverse transcriptase heeft een efficiëntie van reverse transcriptie van 40-60% en de cDNA-opbrengst kan worden verhoogd door een hogere RNA-belading. King reverse transcriptase kan een reverse transcriptie-efficiëntie van meer dan 95% bereiken dankzij de unieke hoge affiniteit voor RNA-templates. Daarom kan aan volgende experimenten worden voldaan zonder dat er een grote hoeveelheid RNA-invoer nodig is, waardoor RNA wordt bespaard en een hoge zuiverheid en een hoge opbrengst aan cDNA mogelijk is.
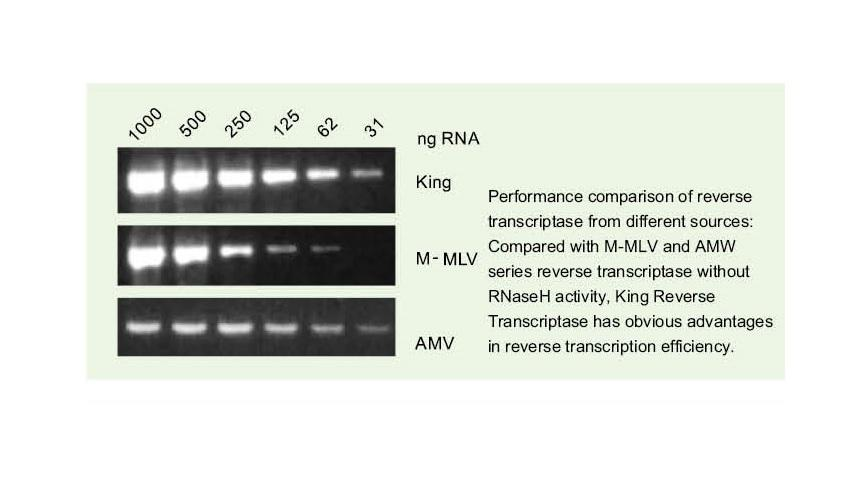
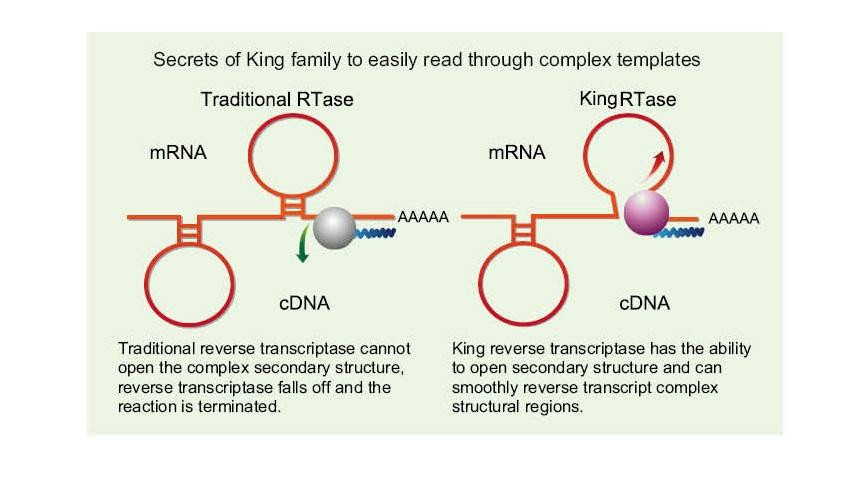
Eenvoudig complexe sjablonen lezen
——Gemakkelijk lezen door hoge GC en complexe sjablonen
Enkelstrengs RNA heeft een breed scala aan complexe secundaire structuurgebieden vanwege waterstofbinding tussen strengen. Gewone reverse transcriptase kan leiden tot beëindiging van reverse transcriptie wanneer een complexe secundaire structuur wordt aangetroffen, waardoor de cDNA-synthese niet met succes kan worden voltooid. De nieuwe generatie King reverse transcriptase heeft echter een uniek structureel domein, dat de waterstofbinding tussen RNA-strengen kan vernietigen, waardoor de complexe secundaire structuur van RNA wordt geopend en de soepele reverse transcriptie wordt gegarandeerd.
Alle producten kunnen worden aangepast voor ODM/OEM. Voor details,klik op Aangepaste Service (ODM/OEM)
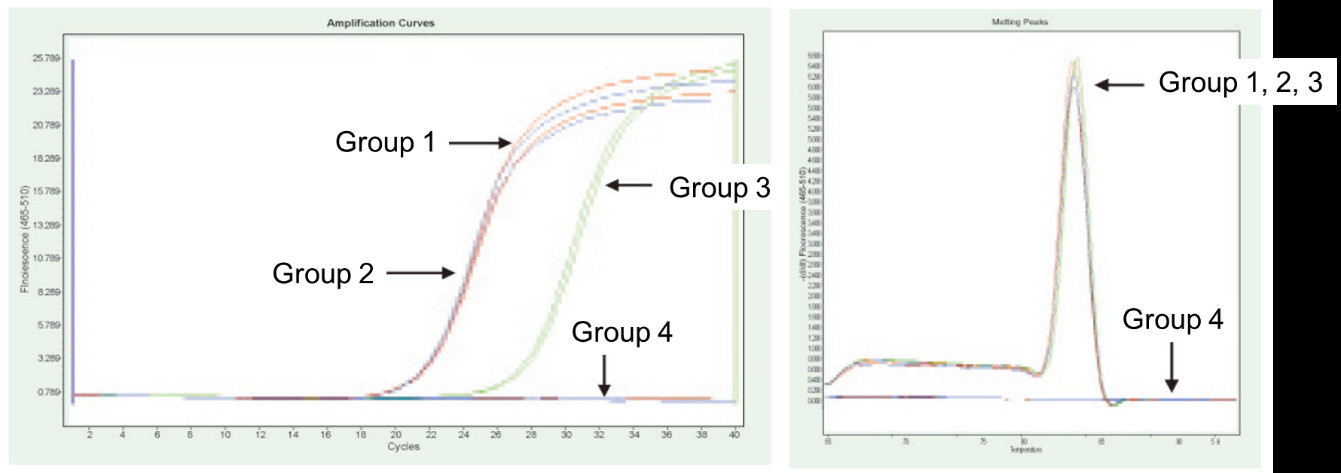 |
Groep 1: Reverse transcriptie zonder gDNase-behandeling; Groep 2: Geen gDNase-behandeling en geen reverse transcriptie; Groep 3: Reverse transcriptie na gDNase-behandeling; Groep 4: gDNase-behandeling zonder reverse transcriptie. Methoden: Fluorescentie kwantitatieve PCR-detectie van het TNF-alfa-gen (primer ontworpen op exon met cDNA of genoom als template) met behulp van 1 μg Hela-cel-RNA (met genoomresidu) als template. Resultaten: Zoals weergegeven in de figuur, kan groep 2 reflecteren het residu van het genoom in RNA, groep 3 kan het ware expressieniveau van TNF-alfa nauwkeurig weergeven, groep 1 heeft fouten in de uiteindelijke kwantitatieve resultaten als gevolg van genoomresidu, en groep 4 laat zien dat FastKing RT Kit het resterende genomische DNA volledig kan verwijderen in RNA. |
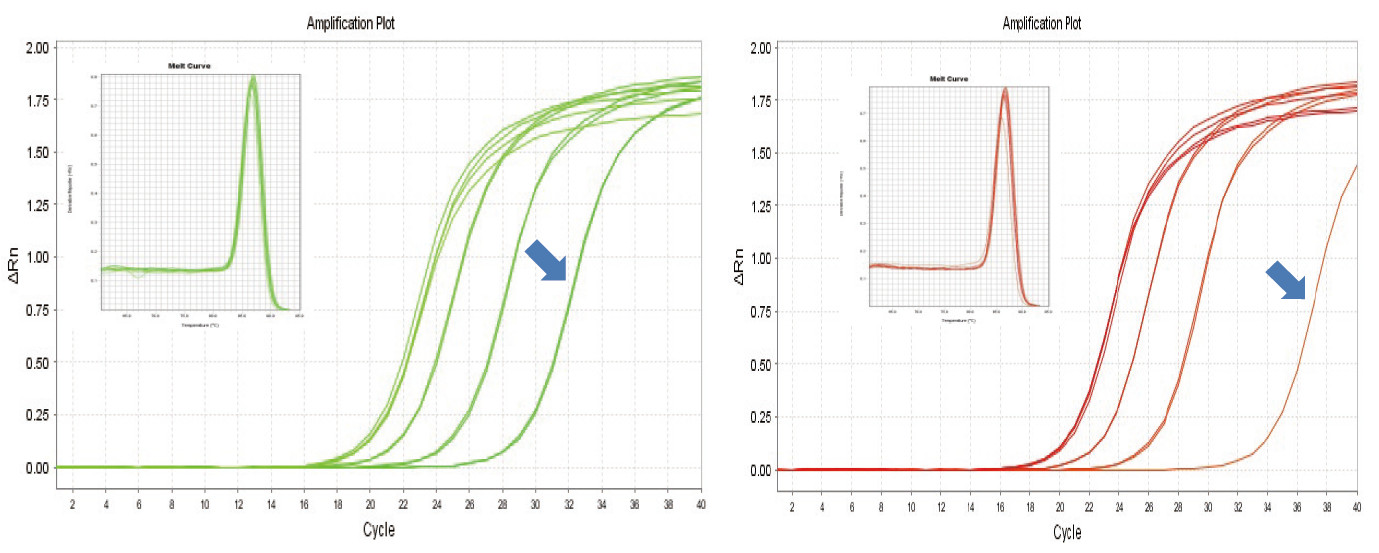 |
Figuur 1. Reverse transcriptie van muis-RNA werd uitgevoerd met behulp van TIANGEN FastKing RT Kit (links) en relevant product van leverancier A (rechts), waarna het MM5-gen kwantitatief werd geamplificeerd met behulp van TIANGEN SuperReal PreMix Plus (SYBR Green). De amplificatiecurve en smeltcurve werden geanalyseerd. De RNA-invoer was respectievelijk 1000 ng, 100 ng, 10 ng en 1 ng. De resultaten laten zien dat TIANGEN FastKing RT Kit een duidelijke omgekeerde transcriptiegradiënt en een lage Ct-waarde heeft, en duidelijke voordelen heeft voor omgekeerde transcriptie van een sjabloon met een lage abundantie (1 ng, blauwe pijl). |
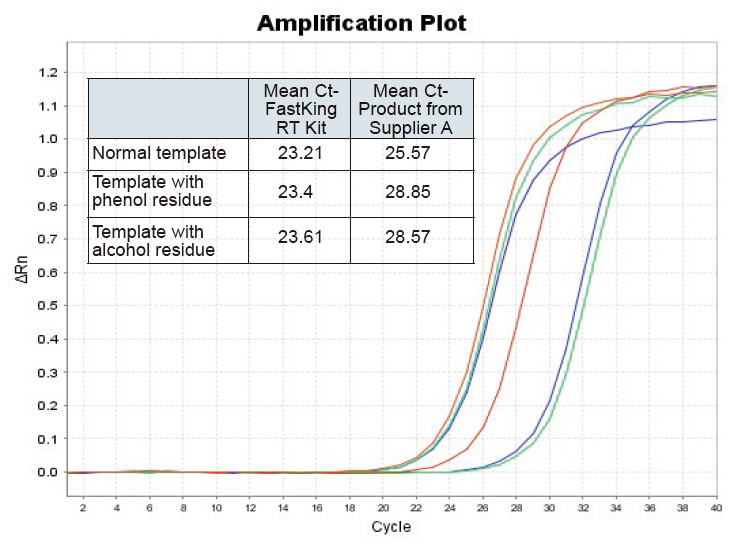 |
Figuur 2. Reverse transcriptie van normaal RNA-template (rood), template met grote fenolrest (groen) en template met alcoholrest (blauw) van ratten met respectievelijk TIANGEN FastKing RT Kit en relevant product van leverancier A, kwantificeer RNC-genen met TIANGEN SuperReal PreMix Plus (SYBR Green), en amplificatiecurven en Ct-waarden werden geanalyseerd. De resultaten tonen aan dat TIANGEN FastKing RT Kit de laagste kwantitatieve Ct-waarde heeft na reverse transcriptie en uitstekende stressbestendigheid, en duidelijke voordelen heeft voor sjablonen met hoge onzuiverheidsresiduen |
A-1 RNA wordt afgebroken
——Zuiver RNA van hoge kwaliteit zonder verontreiniging. Het materiaal waaruit RNA wordt geëxtraheerd, moet zo vers mogelijk zijn om RNA-afbraak te voorkomen. Analyseer RNA-integriteit op gedenatureerde gel vóór RT-reactie. Na RNA-extractie moet het worden bewaard in 100% formamide. Als RNase-remmer wordt gebruikt, moet de verwarmingstemperatuur <45 °C zijn en moet de pH lager zijn dan 8,0, anders zal de remmer alle gebonden RNase afgeven. Bovendien moet RNase-remmer worden toegevoegd in oplossingen die ≥ 0,8 mM DTT bevatten.
A-2 RNA bevat remmers van reverse transcriptiereacties
——Reverse-transcriptieremmers omvatten SDS, EDTA, glycerol, natriumpyrofosfaat, spermidine, formamide, guanidinezout, enz. Meng het controle-RNA met het monster en vergelijk de opbrengst met de controle-RNA-reactie om te controleren of er een remmer is. Was RNA-precipitatie met 70% (v/v) ethanol om remmers te verwijderen.
A-3 Onvoldoende annealing van primers die zijn gebruikt voor het synthetiseren van de eerste cDNA-streng
——Bepaal dat de gloeitemperatuur geschikt is voor de primers die in het experiment worden gebruikt. Voor willekeurige hexameren wordt aanbevolen om de temperatuur gedurende 10 minuten op 25°C te houden voordat de reactietemperatuur wordt bereikt. Probeer voor genspecifieke primers (GSP) een ander GSP of schakel over naar oligo(dT) of willekeurige hexameer.
A-4 Kleine hoeveelheid start-RNA
——Verhoog de hoeveelheid RNA. Voor RNA-monsters van minder dan 50 ng kan 0,1 g tot 0,5 μg acetyl BSA worden gebruikt in de cDNA-synthese van de eerste streng
A-5 De doelsequentie komt niet tot expressie in de geanalyseerde weefsels.
——Probeer andere weefsels.
A-6 PCR-reactie mislukt
——Voor tweestaps RT-PCR mag de cDNA-template in de PCR-stap niet groter zijn dan 1/5 van het reactievolume.
A-1 Niet-specifieke annealing van primers en templates
——Het 3'-uiteinde van primers mag geen 2-3 dG of dC bevatten. Gebruik genspecifieke inleidingen in de eerste strengsynthese in plaats van willekeurige inleidingen of oligo(dT). Gebruik de eerste paar cycli een hogere gloeitemperatuur en daarna een lagere gloeitemperatuur. Gebruik hot-start Taq DNA-polymerase voor PCR om de specificiteit van de reactie te verbeteren.
A-2 Slecht ontwerp van genspecifieke primers
——Volg dezelfde principes voor het ontwerp van de amplificatieprimer.
A-3 RNA besmet met genomisch DNA
——Behandel RNA met PCR-grade DNase I. Stel een controlereactie op zonder reverse transcriptie om DNA-contaminatie te detecteren.
A-4 Vorming van primerdimeer
——Ontwerp primers zonder complementaire sequenties aan het 3'-uiteinde.
A-5 Te hoge Mg2+ concentratie
——Mg . optimaliseren2+ concentratie voor elke combinatie van sjabloon en primer
A-6 Verontreinigd met vreemd DNA
——Gebruik aerosolbestendige tips en UDG-enzymen.
A-1 De inhoud van het eerste strengproduct is te hoog
——Verminder de hoeveelheid van het eerste strengproduct in de conventionele PCR-reactiestap.
A-2 Te hoge hoeveelheid primer in PCR-reactie
——Verminder de invoer van de primer.
A-3 Te veel cycli
— Optimaliseer de PCR-reactieomstandigheden en verlaag het aantal PCR-cycli.
A-4 Te lage gloeitemperatuur
——Verhoog de gloeitemperatuur om niet-specifieke initiatie en verlenging te voorkomen.
A-5 Niet-specifieke amplificatie van oligonucleotidefragmenten gegenereerd door DNase-degradatie van DNA ——Extract RNA van hoge kwaliteit om DNA-contaminatie te voorkomen.
RT-PCR is om RNA omgekeerd te transcriberen in cDNA en vervolgens het omgekeerd getranscribeerde cDNA te gebruiken als een sjabloon voor PCR-reactie om het doelfragment te amplificeren. Kies willekeurige inleidingen, Oligo dT en genspecifieke inleidingen volgens de specifieke voorwaarden van het experiment. Alle bovenstaande primers kunnen worden gebruikt voor kort eukaryotisch cel-mRNA zonder haarspeldstructuur.
Willekeurige primer: Geschikt voor lang RNA met haarspeldstructuur, evenals alle soorten RNA zoals rRNA, mRNA, tRNA, enz. Ze worden voornamelijk gebruikt voor RT-PCR-reactie van een enkele sjabloon.
Oligo dT: Geschikt voor RNA met PolyA-staart (prokaryotisch RNA, eukaryotisch Oligo dT rRNA en tRNA hebben geen PolyA-staarten). Omdat Oligo dT is gebonden aan PolyA-staart, moet de kwaliteit van RNA-monsters hoog zijn, en zelfs een kleine hoeveelheid afbraak zal de hoeveelheid cDNA-synthese van volledige lengte aanzienlijk verminderen.
Genspecifieke primer: complementair aan de templatesequentie, geschikt voor situaties waarbij de targetsequentie bekend is.
Er zijn twee manieren:
1. Interne referentiemethode: in theorie is cDNA DNA-fragmenten van verschillende lengtes, dus het resultaat van elektroforese is uitstrijkje. Als de RNA-abundantie laag is, zal er geen product te zien zijn in elektroforese, maar dit betekent niet dat er geen product zal worden geamplificeerd door PCR. In het algemeen kan interne referentie worden gebruikt om cDNA te detecteren. Als de interne referentie resultaten heeft, kan de kwaliteit van cDNA in principe worden gegarandeerd (in enkele gevallen, als het doelgenfragment te lang is, kunnen er uitzonderingen zijn).
2. Als er een bekend gen is dat door deze matrijs wordt geamplificeerd, kan dit worden geverifieerd door de primers van dit gen. De amplificatie van interne referentie betekent niet noodzakelijk dat er geen probleem is met cDNA. Omdat interne referentie een hoge overvloed aan cDNA heeft, is het gemakkelijk te amplificeren. Als cDNA om verschillende redenen gedeeltelijk wordt afgebroken, zullen de PCR-resultaten van doelwitgenen met een lage abundantie, vanuit het perspectief van waarschijnlijkheid, sterk worden beïnvloed. Hoewel er nog steeds veel interne referentie is, zal de versterking waarschijnlijk niet worden beïnvloed.
Gedeeltelijk degraderen van RNA. Detecteer de integriteit en zuiveren van RNA
De RNA-inhoud van verschillende soorten kan verschillen, maar in het algemeen moet het geëxtraheerde totale RNA twee duidelijke 28S- en 18S-banden bevatten bij gelelektroforese, en de helderheid van de eerste band moet twee keer zo hoog zijn als die van de laatste. De 5S-band geeft aan dat RNA is afgebroken en de helderheid ervan is evenredig met de mate van afbraak. De succesvolle amplificatie van interne referentie betekent niet dat er geen probleem is met RNA, omdat de interne referentie in grote hoeveelheden aanwezig is, RNA kan worden geamplificeerd zolang de afbraak niet ernstig is. de OD260/OD280verhouding van puur RNA gemeten met een spectrofotometer moet tussen 1,9 en 2,1 liggen. Een kleine hoeveelheid eiwitonzuiverheid in RNA zal de verhouding verminderen. Zolang de waarde niet te laag is, wordt de RT niet beïnvloed. Het belangrijkste voor RT is de RNA-integriteit.
De verlenging van het interne referentiegen kan alleen maar aangeven dat RT is geslaagd, maar is niet noodzakelijk gerelateerd aan de kwaliteit van de cDNA-streng. Omdat de interne referentiefragmenten over het algemeen klein van formaat en hoog in expressie zijn, zijn ze gemakkelijker succesvol in reverse transcriptie. De grootte en expressie van het doelgen varieert echter van gen tot gen. De cDNA-kwaliteit kan niet alleen worden beoordeeld door interne referentie, vooral voor de doelfragmenten die langer zijn dan 2 kb.
Sommige monsters hebben complexe secundaire structuren, of hebben een rijk GC-gehalte, of zijn kostbaar met een lage abundantie. In deze gevallen moet de juiste reverse transcriptase worden geselecteerd op basis van de grootte van het doelfragment en het monster. Voor RNA-templates met een hoog GC-gehalte en een complexe secundaire structuur is het moeilijk om de secundaire structuur te openen bij lage temperatuur of met gewone reverse transcriptase. Voor deze sjablonen kan Quant Reverse Transcriptase worden geselecteerd, aangezien de prestaties van reverse transcriptie duidelijk beter zijn dan die van reverse transcriptase uit de M-MLV-serie, die verschillende RNA-templates efficiënt kan reverse transcriberen en RNA maximaal in de eerste cDNA-streng kan transcriberen. Bij gebruik van een algemene reverse transcriptase-kit, kan het 20 l-systeem slechts 1 g totaal RNA effectief reverse transcriberen. Let op de maximale RT-capaciteit van de kit. Als de sjabloon in overmaat wordt toegevoegd, zal reverse transcriptie het RNA met een hoge overvloed bevoordelen. Daarom is het beter om de maximale capaciteit van het systeem niet te overschrijden.
A-1 Bepaal of RNA ernstig is afgebroken en of RT succesvol is
Over het algemeen wordt de reden voor het falen van interne referentieamplificatie vaak veroorzaakt door ernstige RNA-afbraak. Een andere mogelijke reden is het falen van de reverse transcriptie. Interne referentie kan niet als standaard worden gebruikt om de kwaliteit van enkelstrengs cDNA te beoordelen, maar kan wel als standaard worden gebruikt om te beoordelen of reverse transcriptie succesvol is als er geen probleem is met de RNA-kwaliteit. Het belangrijkste in het reverse transcriptieproces is het handhaven van een constante temperatuur en een constant reactiesysteem om de reactie-efficiëntie te verbeteren.
A-2 Bepaal of de primers voor het amplificeren van interne referentiegenen betrouwbaar zijn en of er problemen zijn met de reagentia die bij PCR worden gebruikt.
Voor relatieve kwantificering moet RNA worden gekwantificeerd vóór reverse transcriptie, wat ook vereist is in veel reverse transcriptiekits, bijvoorbeeld de RNA-invoer kwantificeren als 1 μg. Omdat het omgekeerd getranscribeerde cDNA een gemengde oplossing is, inclusief RNA, oligo-dT, enzym, dNTP en zelfs een beetje DNA-residu, zal er een afwijking optreden, dus het is onmogelijk om het cDNA nauwkeurig te kwantificeren. Daarom is RNA-kwantificering noodzakelijk. Aangezien de efficiëntie van omgekeerde transcriptie hetzelfde is tussen verschillende monsters, moet de verkregen hoeveelheid cDNA hetzelfde zijn en kan de kwantitatieve analyse de vergelijking van expressieniveaus van verschillende genen in dezelfde hoeveelheid totaal RNA laten zien. Bij het uitvoeren van kwantitatieve PCR met relatieve fluorescentie, is kwantitatief cDNA mogelijk niet vereist na reverse transcriptie omdat het interne referentiegen als referentie kan dienen.
Het is voornamelijk gerelateerd aan de genen en reverse transcriptie van een lang fragment is voor de meeste genen niet haalbaar. Ten eerste is de efficiëntie van reverse transcriptie veel lager dan die van PCR. Ten tweede beperken het GC-rijke gebied en de secundaire structuur van veel genen zowel reverse transcriptie als PCR. Ten slotte zijn de betrouwbaarheid en versterkingsefficiëntie van PCR moeilijk tegelijkertijd te garanderen. In het proces van reverse transcriptie kan niemand garanderen dat een lang fragment wordt verkregen voor genen met een lage kopie, vooral met behulp van oligo dT. Wat betreft 5' UTR met meer GC, het is nog moeilijker. Daarom is het nog steeds een redelijke methode om transcript met willekeurige primers om te keren, de natuurlijke splitsingsplaatsen in het doelfragment te vinden, door segmenten te amplificeren en vervolgens de restrictiedigestie en ligatie uit te voeren. In het algemeen is het moeilijk om fragmenten groter dan 2 kb direct te amplificeren, maar het is niet altijd onmogelijk om: 1. Allereerst de integriteit van RNA/mRNA garanderen, en TRIZOL-extractie heeft de voorkeur. 2.M-MLV RT-PCR-kit kan direct worden gebruikt. Verleng de gloeitijd en verhoog het aantal cycli in het amplificatieproces op de juiste manier. Als alternatief kan geneste PCR worden toegepast, of eerst een of twee reacties uitvoeren met de juiste langere denaturatie en verlengingstijd vóór normale PCR-amplificatie, wat kan helpen om fragmenten te verlengen. Besteed aandacht aan de getrouwheid van het polymerase. 3.Long Taq kan in PCR worden gebruikt om ideale resultaten te verkrijgen. 4. Voor toepassing van eiwitexpressie moet high-fidelity-polymerase worden toegepast.
Er zijn twee soorten reverse transcriptase aangeboden door TIANGEN: Quant/King RTase en TIANScript M-MLV. Het belangrijkste verschil tussen hen is het aantal ingevoerde sjablonen. Quant is een unieke reverse transcriptase, die verschilt van de veelgebruikte M-MLV die is afgeleid van het Moloney-muizenleukemievirus. Quant is een nieuwe zeer efficiënte reverse transcriptase die op recombinante wijze tot expressie wordt gebracht door engineering van Escherichia coli. Quant is geschikt voor het amplificeren van 50 ng-2 μg RNA met hoge reverse transcriptionele activiteit en hoge opbrengst. Vergeleken met gewone MMLV of AMV is Quants grootste kenmerk dat het een zeer sterke affiniteit heeft met RNA-templates en transcript-complextemplates kan omkeren zonder denaturatie bij hoge temperatuur. Voor sjablonen met een hoger GC-gehalte is de omgekeerde efficiëntie hoger. Deze reverse transcriptase heeft echter RNase H-activiteit, wat de lengte van het cDNA-product kan beïnvloeden (geschikt voor templates van < 4,5 kb). Voor conventionele reverse transcriptie wordt TIANScript MMLV reverse transcriptase aanbevolen. Dit RTase is een gemodificeerd enzym met een zeer zwakke RNase H-activiteit, dat geschikt is voor lange (> 5 kb) cDNA-synthese.
Eenstaps reverse transcriptie en PCR-amplificatie worden in dezelfde buis voltooid zonder de buisafdekking tussen cDNA-synthese en amplificatie te openen, wat nuttig is om contaminatie te verminderen. Omdat alle verkregen cDNA-monsters worden gebruikt voor amplificatie, is de gevoeligheid hoger, met een minimum van 0,01 pg totaal RNA. Voor succesvolle eenstaps RTPCR worden over het algemeen genspecifieke primers gebruikt om cDNA-synthese te initiëren. De tweestapsmethode, namelijk reverse transcriptie en PCR-amplificatie, wordt in twee stappen uitgevoerd. Ten eerste wordt reverse transcriptie uitgevoerd vanaf een RNA-matrijs om cDNA te verkrijgen, en het verkregen cDNA wordt onderworpen aan een of meer verschillende PCR-reacties. De tweestapsmethode kan oligo(dT) of willekeurige primers gebruiken om de synthese van de eerste cDNA-streng te begeleiden, en kan alle mRNA-informatie van een specifiek monster omgekeerd transcriberen.
Producten categorieën
WAAROM VOOR ONS KIEZEN
Sinds de oprichting heeft onze fabriek eersteklas producten ontwikkeld volgens het principe:
van kwaliteit voorop. Onze producten hebben een uitstekende reputatie verworven in de industrie en zijn waardevol bij nieuwe en oude klanten.










