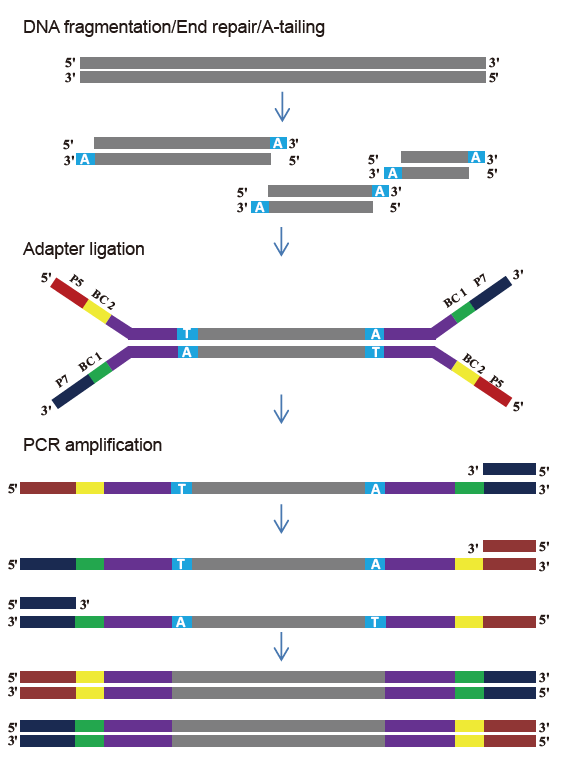
TIANSeq DirectFast bibliotheekkit (illumina)
Functies
■ Goede sequentie-uniformiteit: geen basenvertekening van het DNA-fragmentatieproces en PCR-amplificatieproces.
■ Hoge bibliotheekconversie-efficiëntie: de zeer efficiënte bibliotheekconstructie kan worden gegarandeerd voor 1 ng DNA-monsters.
■ Snelle bediening: het hele bouwproces van de bibliotheek duurt slechts 2,5 uur.
■ Kostenefficiënt: er zijn geen speciale instrumenten en apparatuur nodig。
Specificatie
Type: DNA-bibliotheekvoorbereiding voor illumina high-throughput sequencing-platform
Steekproef: Genomisch DNA of groot fragment-DNA
Doelwit: Dubbelstrengs DNA
Voorbeeldinvoer starten: 1 ng- 1 g
Operatie tijd: 2,5 uur
Downstream-toepassingen: Sequencing op illumina platform
Alle producten kunnen worden aangepast voor ODM/OEM. Voor details,klik op Aangepaste Service (ODM/OEM)
Flexibele monsterinvoer en gefragmenteerde grootte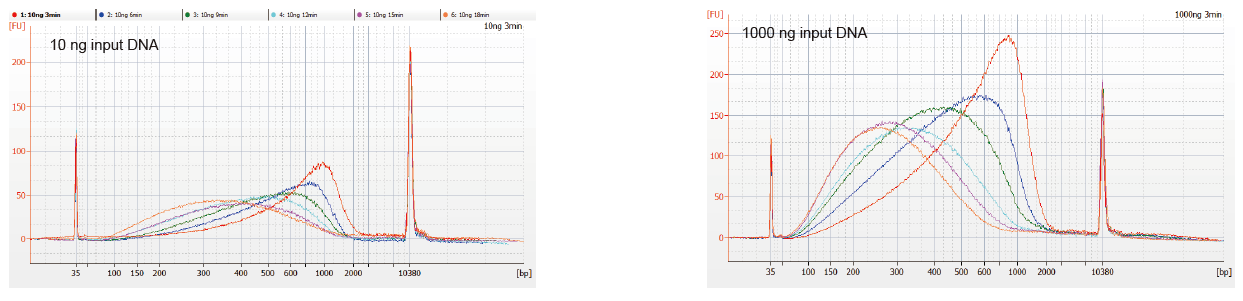 |
Figuur 1. DNA-fragmentatieprofielen van verschillende reactietijden. 10 ng en 1000 ng DNA werden gefragmenteerd met behulp van TIANSeq DirectFast DNA Library Kit. De met verschillende reactietijden behandelde reactieproducten werden gezuiverd door 1,8 x Ampure XP magnetische korrels en geanalyseerd door Angilent 2100. |
Covaris-achtige sequentiedekking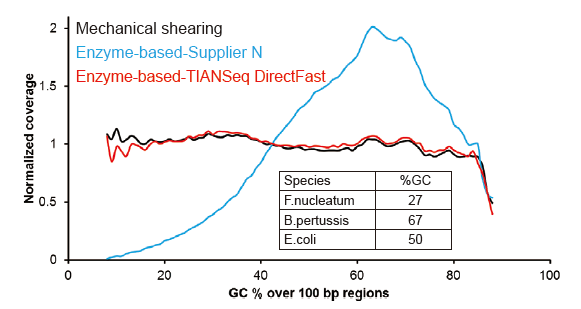 |
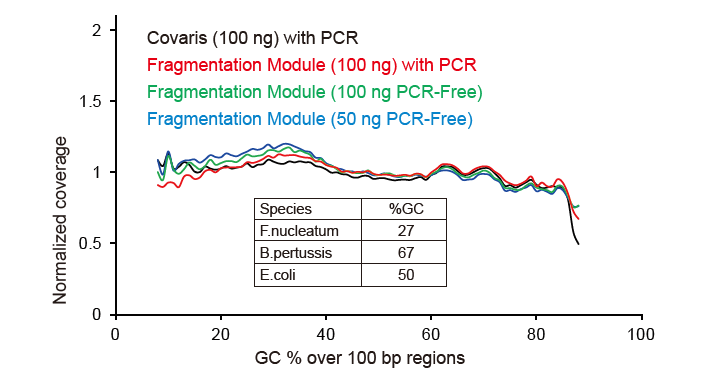
Figuur 2. Vergelijking van genoomdekking van verschillende bibliotheekvoorbereidingsmethoden. Drie bacterieel genomisch DNA met verschillende GC-inhouden zijn equimolair gemengd en het sequentie-genoomdekkingsresultaat van 100 ng gemengde DNA-bibliotheken met behulp van deze methoden werd vergeleken. De resultaten laten zien dat de TIANSeq DirectFast Library Kit hetzelfde effect heeft op DNA-fragmentatie als mechanische shearing, en dat er geen base-bias is voor fragmentatie. |
Geen systematische bias voor slechts 1 ng input-DNA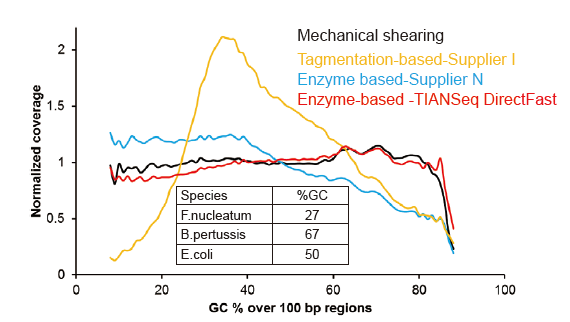 |
Figuur 3. Vergelijking van genoomdekking van verschillende bibliotheekvoorbereidingsmethoden. Drie bacterieel genomisch DNA met verschillende GC-inhouden zijn equimolair gemengd en het sequentie-genoomdekkingsresultaat van 1 ng gemengde DNA-bibliotheken met behulp van deze methoden werd vergeleken. De resultaten laten zien dat de TIANSeq DirectFast Library Kit een consistent fragmentatie-effect heeft met de mechanische afschuiving, zelfs voor DNA-invoer van slechts 1 ng, en er is geen base-bias. |
| Geschikt voor PCR-vrije workflow
|
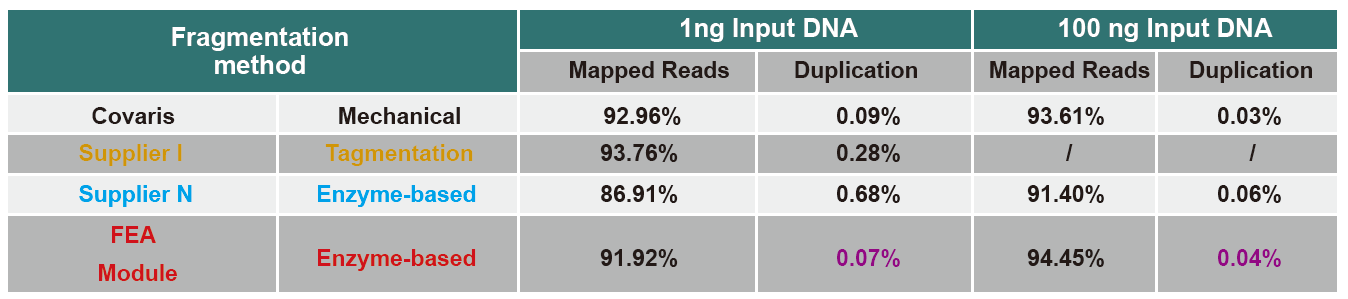
Figuur 4. Verschillende input van genomisch DNA werd gebruikt om de bibliotheek te construeren door middel van PCR of PCR-vrije bibliotheekconstructie, en de resultaten van de genoomdekking werden vergeleken. De resultaten laten zien dat met de werking met één buis en efficiënte bibliotheekconstructiestappen, de DNA-bibliotheek die is gebouwd met TIANSeq DirectFast Library Kit een hoge consistentie behoudt met de mechanische afschuiving in de distributie van fragmentsequentiedekking voor zowel PCR-verrijking PCR-vrije workflow. |
Statistieken van de efficiëntie en opbrengst van bibliotheekconstructies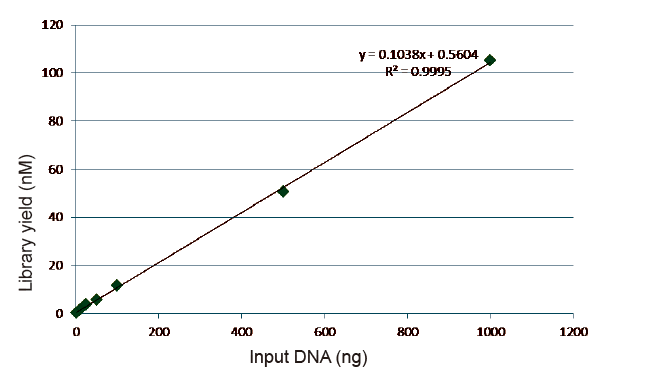 |
Figuur 5. Resultaten van kwantitatieve analyse van bibliotheek-DNA verkregen door qPCR na bibliotheekconstructie door PCR-vrije methode voor monsters met verschillende startbedragen (1, 10, 25, 50, 100, 500,1000 ng). Lineaire regressieanalyse laat zien dat de bibliotheekopbrengst een goede lineaire relatie heeft in een breed invoerbereik voor monsters. Voor DNA-invoer van slechts 1 ng neemt de efficiëntie van de bibliotheekconstructie niet af. |
Vergelijking van sequentiegegevens van verschillende producten
Op dit moment is high-throughput sequencing-technologie voornamelijk gebaseerd op de volgende generatie sequencing-technologie. Omdat de leeslengte van de volgende generatie sequencing-technologie beperkt is, moeten we de sequentie van volledige lengte opsplitsen in kleine fragmentbibliotheken om te sequensen. Afhankelijk van de behoeften van verschillende sequencing-experimenten, kiezen we meestal voor single-ended sequencing of double-ended sequencing. Momenteel zijn de DNA-fragmenten van de volgende generatie sequencing-bibliotheek over het algemeen verdeeld in het bereik van 200-800 bp.

a) DNA is van slechte kwaliteit en bevat remmers. Gebruik hoogwaardige DNA-monsters om remming van enzymactiviteit te voorkomen.
b) De hoeveelheid DNA-monster is onvoldoende bij gebruik van een PCR-vrije methode om de DNA-bibliotheek te construeren. Wanneer de invoer van het gefragmenteerde DNA 50 ng overschrijdt, kan een PCR-vrije workflow selectief worden uitgevoerd tijdens het bouwproces van de bibliotheek. Als het aantal kopieën van de bibliotheek te laag is om direct te worden gesequenced, kan de DNA-bibliotheek worden geamplificeerd door PCR na de adapterligatie.
c) RNA-contaminatie leidt tot onnauwkeurige initiële DNA-kwantificering RNA-contaminatie kan voorkomen in het zuiveringsproces van genomisch DNA, wat kan leiden tot onnauwkeurige DNA-kwantificering en onvoldoende DNA-lading tijdens bibliotheekconstructie. RNA kan worden verwijderd door te behandelen met RNase.
A-1
a) Kleine fragmenten (60 bp-120 bp) verschijnen Kleine fragmenten zijn gewoonlijk adapterfragmenten of dimeren gevormd door adapters. Zuivering met magnetische korrels van Agencourt AMPure XP kan deze adapterfragmenten effectief verwijderen en de kwaliteit van de sequentie garanderen.
b) Grote fragmenten verschijnen in de bibliotheek na PCR-amplificatie. De grootte van het DNA-fragment van de bibliotheek zal met 120 bp toenemen nadat de adapter is geligeerd. Als het DNA-fragment na de adapterligatie met meer dan 120 bp toeneemt, kan dit worden veroorzaakt door abnormale fragmentamplificatie of overmatige PCR-amplificatie. Het verminderen van het aantal PCR-cycli kan de situatie voorkomen.
c) Abnormale grootte van bibliotheek-DNA-fragmenten na adapterligatie De lengte van de adapter in deze kit is 60 bp. Wanneer de twee uiteinden van het fragment aan de adapters worden geligeerd, neemt de lengte slechts met 120 bp toe. Als u een andere adapter gebruikt dan de adapter die in deze kit wordt geleverd, neem dan contact op met de leverancier om relevante informatie te verstrekken, zoals de lengte van de adapter. Zorg ervoor dat de werkstroom en werking van het experiment de stappen volgen die in de handleiding worden beschreven.
d) Abnormale DNA-fragmentgrootte vóór de adapterligatie De reden voor dit probleem kan worden veroorzaakt door verkeerde reactieomstandigheden tijdens DNA-fragmentatie. Er moeten verschillende reactietijden worden gebruikt voor verschillende DNA-invoer. Als de DNA-invoer meer dan 10 ng is, raden we aan om de reactietijd van 12 min te kiezen als starttijd voor optimalisatie, en de fragmentgrootte die op dit moment wordt geproduceerd, ligt voornamelijk in het bereik van 300-500 bp. Gebruikers kunnen de lengte van DNA-fragmenten gedurende 2-4 minuten volgens hun eigen vereisten vergroten of verkleinen om de DNA-fragmenten met de vereiste grootte te optimaliseren.
A-2
a) Fragmentatietijd is niet geoptimaliseerd Als het gefragmenteerde DNA te klein of te groot is, raadpleeg dan de richtlijnen voor selectie van fragmentatietijd in de instructie om de reactietijd te bepalen, en gebruik dit tijdstip als controle, stel daarnaast een reactiesysteem om 3 min te verlengen of te verkorten om een nauwkeurigere aanpassing te maken aan de fragmentatietijd.
A-3
Abnormale grootteverdeling van DNA na fragmentatiebehandeling
a) Onjuiste ontdooimethode van fragmentatiereagens, of het reagens is niet volledig gemengd na ontdooien. Ontdooi het 5× Fragmentation Enzyme Mix-reagens op ijs. Eenmaal ontdooid, mengt u het reagens gelijkmatig door zachtjes tegen de onderkant van de buis te tikken. Vortex het reagens niet!
b) Het DNA-invoermonster bevat EDTA of andere verontreinigende stoffen. Uitputting van zoutionen en chelaatvormers in de DNA-zuiveringsstap is bijzonder belangrijk voor het succes van het experiment. Als DNA is opgelost in 1×TE, gebruik dan de methode die in de instructie wordt gegeven om fragmentatie uit te voeren. Als de EDTA-concentratie in de oplossing onzeker is, wordt aanbevolen om het DNA te zuiveren en op te lossen in gedeïoniseerd water voor een daaropvolgende reactie.
c) Onnauwkeurige initiële DNA-kwantificering De grootte van gefragmenteerd DNA hangt nauw samen met de hoeveelheid DNA-invoer. Vóór de fragmentatiebehandeling is nauwkeurige kwantificering van DNA met behulp van Qubit, Picogreen en andere methoden essentieel om de exacte hoeveelheid DNA in het reactiesysteem te bepalen.
d) De bereiding van het reactiesysteem volgt niet de instructie. De bereiding van het gefragmenteerde reactiesysteem moet strikt volgens de instructies op ijs worden uitgevoerd. Om het beste effect te garanderen, moeten alle reactiecomponenten op ijs worden geplaatst en moet de voorbereiding van het reactiesysteem worden uitgevoerd na volledige afkoeling. Nadat de bereiding is voltooid, kunt u tikken of pipetteren om grondig te mengen. Niet vortexen!
1. Onjuiste mengmethode (vortex, hevige oscillatie, enz.) zal een abnormale verdeling van bibliotheekfragmenten veroorzaken (zoals weergegeven in de volgende afbeelding), waardoor de kwaliteit van de bibliotheek wordt aangetast. Daarom moet u bij het bereiden van de Fragmentation Mix-reactieoplossing voorzichtig op en neer pipetteren om te mengen, of gebruik de vingertop om gelijkmatig te bewegen en te mengen. Wees voorzichtig niet te mengen met vortex.

2. DNA van hoge zuiverheid moet worden gebruikt voor bibliotheekconstructie
■ Goede DNA-integriteit: de elektroforeseband is meer dan 30 kb, zonder tailing
■ OD260/230: >1,5
■ OD260/280: 1,7-1,9
3. Hoeveelheid DNA-invoer moet nauwkeurig zijn. Er wordt voorgesteld om Qubit- en PicoGreen-methoden te gebruiken om DNA te kwantificeren in plaats van Nanodrop.
4. Het gehalte aan EDTA in DNA-oplossing moet worden bepaald. EDTA heeft een grote invloed op de fragmentatiereactie. Als het EDTA-gehalte hoog is, moet vóór de volgende test DNA-zuivering worden uitgevoerd.
5. De fragmentatiereactieoplossing moet op ijs worden bereid. Het fragmentatieproces is gevoelig voor reactietemperatuur en -tijd (vooral na toevoeging van versterker). Bereid het reactiesysteem op ijs voor om de nauwkeurigheid van de reactietijd te garanderen.
6. De reactietijd van de fragmentatie moet nauwkeurig zijn. De reactietijd van de fragmentatiestap heeft direct invloed op de grootte van de fragmentproducten, en dus op de grootteverdeling van DNA-fragmenten in de bibliotheek.
1. Welk type monster is van toepassing op deze kit?
Het toepasselijke monstertype van deze kit kan totaal RNA of gezuiverd mRNA zijn met een goede RNA-integriteit. Als totaal RNA wordt gebruikt om de bibliotheek te construeren, wordt aanbevolen om de rRNA-depletiekit (Cat #4992363/4992364/4992391) te gebruiken om eerst rRNA te verwijderen.
2. Kunnen FFPE-samples worden gebruikt om een bibliotheek te bouwen met deze kit?
Het mRNA in FFPE-monsters zal tot op zekere hoogte worden afgebroken, met een relatief slechte integriteit. Bij gebruik van deze kit voor de bibliotheekconstructie, wordt aanbevolen om de fragmentatietijd te optimaliseren (verkort de fragmentatietijd of voer geen fragmentatie uit).
3. Wat kan ervoor zorgen dat het ingevoegde segment een kleine afwijking lijkt te hebben, met behulp van de stap voor het selecteren van de maat in de producthandleiding?
De maatkeuze wordt uitgevoerd in strikte overeenstemming met de maatkeuzestap in deze producthandleiding. Als er een afwijking is, kan de reden zijn dat de magnetische kralen niet op kamertemperatuur zijn uitgebalanceerd of niet volledig zijn gemengd, de pipet niet nauwkeurig is of de vloeistof in de punt is achtergebleven. Het wordt aanbevolen om de tips met lage adsorptie voor het experiment te gebruiken.
4. Selectie van adapters in bibliotheekconstructie
De bibliotheekconstructiekit bevat geen adapterreagens en het wordt aanbevolen om deze kit samen met de TIANSeq Single-Index Adapter (Illumina) (4992641/4992642/4992378) te gebruiken.
5. QC van de bibliotheek
Bibliotheek kwantitatieve detectie: Qubit en qPCR worden gebruikt om respectievelijk de massaconcentratie en molaire concentratie van de bibliotheek te bepalen. De bediening is strikt in overeenstemming met de producthandleiding. De concentratie van de bibliotheek zal over het algemeen voldoen aan de vereisten van NGS-sequencing. Detectie van bibliotheekdistributiebereik: Agilent 2100 Bioanalyzer gebruiken om het bibliotheekdistributiebereik te detecteren.
6. Selectie van versterkingscyclusnummer:
Volgens de instructies is het aantal PCR-cycli 6-12 en moet het aantal benodigde PCR-cycli worden geselecteerd op basis van de invoer van het monster. In high-yieldbibliotheken treedt overamplificatie meestal in verschillende mate op, wat zich manifesteert door een iets grotere piek na de piek van het doelbereik bij de detectie van Agilent 2100 Bioanalyzer, of de gedetecteerde concentratie van Qubit is lager dan die van qPCR. Milde overversterking is een normaal verschijnsel, dat geen invloed heeft op de bibliotheeksequencing en daaropvolgende gegevensanalyse.
7. Spikes verschijnt in het detectieprofiel van Agilent 2100 Bioanalyzer
Het optreden van pieken in de Agilent 2100 Bioanalyzer-detectie is vanwege de ongelijke fragmentatie van monsters, waarbij er meer fragmenten van een bepaalde grootte zullen zijn, en dit zal duidelijker worden na PCR-verrijking. In dit geval wordt voorgesteld om de grootteselectie niet uit te voeren, dwz de fragmentatieconditie in te stellen op 94°C gedurende 15 minuten geïncubeerd, waar de fragmentverdeling klein en geconcentreerd is en de homogeniteit kan worden verbeterd.
Producten categorieën
WAAROM VOOR ONS KIEZEN
Sinds de oprichting heeft onze fabriek eersteklas producten ontwikkeld volgens het principe:
van kwaliteit voorop. Onze producten hebben een uitstekende reputatie verworven in de industrie en zijn waardevol bij nieuwe en oude klanten.